Rでlmer関数を使って線形混合モデルを作成する
パッケージの読み込み
lmer関数はlme4パッケージに入っています。 まだインストールが済んでいなかったらinstall.packages関数でインストールします。
その後、library関数でパッケージを読み込みます。
使うデータセット
パッケージに入っている、sleepstudyというデータセットが練習として使えそうです。 パッケージを読み込んだ後であれば、そのままデータセット名で実行するだけで読み込めます。
Reaction Days Subject
1 249.5600 0 308
2 258.7047 1 308
3 250.8006 2 308
4 321.4398 3 308
5 356.8519 4 308
6 414.6901 5 308
7 382.2038 6 308
8 290.1486 7 308
9 430.5853 8 308
10 466.3535 9 308
11 222.7339 0 309
12 205.2658 1 309
13 202.9778 2 309
14 204.7070 3 309
15 207.7161 4 309
16 215.9618 5 309
17 213.6303 6 309
18 217.7272 7 309
19 224.2957 8 309
20 237.3142 9 309
21 199.0539 0 310
22 194.3322 1 310
23 234.3200 2 310
24 232.8416 3 310
25 229.3074 4 310
26 220.4579 5 310
27 235.4208 6 310
28 255.7511 7 310
29 261.0125 8 310
30 247.5153 9 310
31 321.5426 0 330
32 300.4002 1 330
33 283.8565 2 330
34 285.1330 3 330
35 285.7973 4 330
36 297.5855 5 330
37 280.2396 6 330
38 318.2613 7 330
39 305.3495 8 330
40 354.0487 9 330
41 287.6079 0 331
42 285.0000 1 331
43 301.8206 2 331
44 320.1153 3 331
45 316.2773 4 331
46 293.3187 5 331
47 290.0750 6 331
48 334.8177 7 331
49 293.7469 8 331
50 371.5811 9 331
51 234.8606 0 332
52 242.8118 1 332
53 272.9613 2 332
54 309.7688 3 332
55 317.4629 4 332
56 309.9976 5 332
57 454.1619 6 332
58 346.8311 7 332
59 330.3003 8 332
60 253.8644 9 332
61 283.8424 0 333
62 289.5550 1 333
63 276.7693 2 333
64 299.8097 3 333
65 297.1710 4 333
66 338.1665 5 333
67 332.0265 6 333
68 348.8399 7 333
69 333.3600 8 333
70 362.0428 9 333
71 265.4731 0 334
72 276.2012 1 334
73 243.3647 2 334
74 254.6723 3 334
75 279.0244 4 334
76 284.1912 5 334
77 305.5248 6 334
78 331.5229 7 334
79 335.7469 8 334
80 377.2990 9 334
81 241.6083 0 335
82 273.9472 1 335
83 254.4907 2 335
84 270.8021 3 335
85 251.4519 4 335
86 254.6362 5 335
87 245.4523 6 335
88 235.3110 7 335
89 235.7541 8 335
90 237.2466 9 335
91 312.3666 0 337
92 313.8058 1 337
93 291.6112 2 337
94 346.1222 3 337
95 365.7324 4 337
96 391.8385 5 337
97 404.2601 6 337
98 416.6923 7 337
99 455.8643 8 337
100 458.9167 9 337
101 236.1032 0 349
102 230.3167 1 349
103 238.9256 2 349
104 254.9220 3 349
105 250.7103 4 349
106 269.7744 5 349
107 281.5648 6 349
108 308.1020 7 349
109 336.2806 8 349
110 351.6451 9 349
111 256.2968 0 350
112 243.4543 1 350
113 256.2046 2 350
114 255.5271 3 350
115 268.9165 4 350
116 329.7247 5 350
117 379.4445 6 350
118 362.9184 7 350
119 394.4872 8 350
120 389.0527 9 350
121 250.5265 0 351
122 300.0576 1 351
123 269.8939 2 351
124 280.5891 3 351
125 271.8274 4 351
126 304.6336 5 351
127 287.7466 6 351
128 266.5955 7 351
129 321.5418 8 351
130 347.5655 9 351
131 221.6771 0 352
132 298.1939 1 352
133 326.8785 2 352
134 346.8555 3 352
135 348.7402 4 352
136 352.8287 5 352
137 354.4266 6 352
138 360.4326 7 352
139 375.6406 8 352
140 388.5417 9 352
141 271.9235 0 369
142 268.4369 1 369
143 257.2424 2 369
144 277.6566 3 369
145 314.8222 4 369
146 317.2135 5 369
147 298.1353 6 369
148 348.1229 7 369
149 340.2800 8 369
150 366.5131 9 369
151 225.2640 0 370
152 234.5235 1 370
153 238.9008 2 370
154 240.4730 3 370
155 267.5373 4 370
156 344.1937 5 370
157 281.1481 6 370
158 347.5855 7 370
159 365.1630 8 370
160 372.2288 9 370
161 269.8804 0 371
162 272.4428 1 371
163 277.8989 2 371
164 281.7895 3 371
165 279.1705 4 371
166 284.5120 5 371
167 259.2658 6 371
168 304.6306 7 371
169 350.7807 8 371
170 369.4692 9 371
171 269.4117 0 372
172 273.4740 1 372
173 297.5968 2 372
174 310.6316 3 372
175 287.1726 4 372
176 329.6076 5 372
177 334.4818 6 372
178 343.2199 7 372
179 369.1417 8 372
180 364.1236 9 372180行3列のデータフレームです。 説明によると、「睡眠不足研究における応答時間」のデータということです。 どうやら、被験者を睡眠不足にして、それによって何かへの応答(反応)速度がどう変わるかを見ているようです。 * Reaction: 平均応答時間 (ms) * Days: 睡眠不足の日数 * Subject: 被験者番号 (18人いる)
str関数を用いることで構造が簡単にわかります。
データの可視化
とりあえず睡眠不足の日数に対して応答速度がどのように変化するかプロットします。 色分けは被験者番号ごとにします。
plot(sleepstudy$Days, sleepstudy$Reaction, col = sleepstudy$Subject, xlab = "Days", ylab = "Reaction Time (ms)", yaxt = "n")
axis(2, las = 2)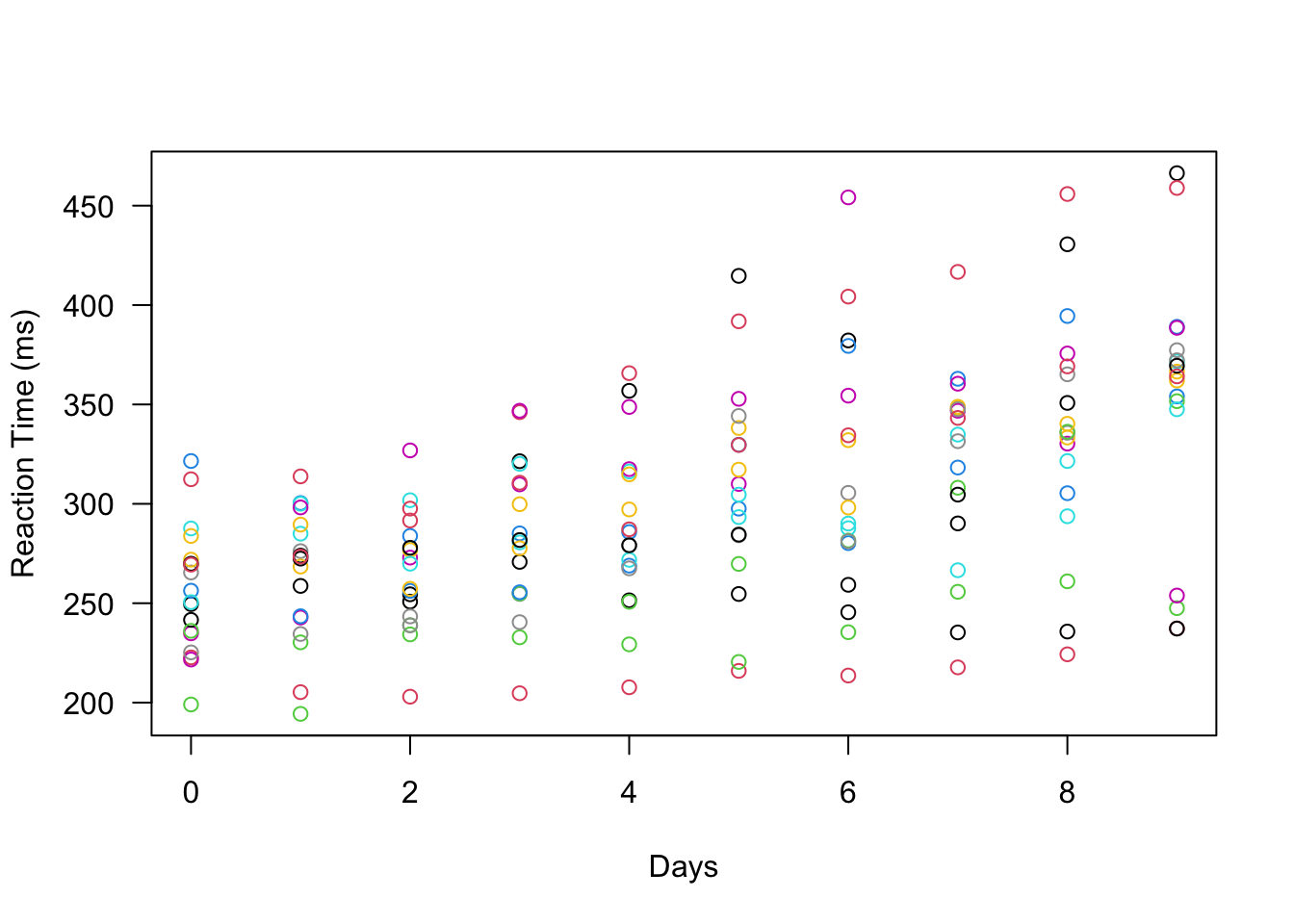
被験者ごとに傾きや切片が異なっていそうとわかります。
線形混合モデルの作成
被験者ごとに傾きや切片が異なっているということで、被験者をランダム効果に入れることで個人差の影響と全体の傾向を分けることができます。 睡眠不足の日数に対し、スタート時点での応答速度(切片)と睡眠不足の影響(傾き)がそれぞれ人によって異なるとすると、(Days | Subject)とすれば切片と傾きをランダム効果にいれることができるようです。 lmer関数でモデルを作成し、そのモデルをsummary関数にいれることでわかりやすく結果を得ることができます。
Linear mixed model fit by REML ['lmerMod']
Formula: Reaction ~ Days + (Days | Subject)
Data: sleepstudy
REML criterion at convergence: 1743.6
Scaled residuals:
Min 1Q Median 3Q Max
-3.9536 -0.4634 0.0231 0.4634 5.1793
Random effects:
Groups Name Variance Std.Dev. Corr
Subject (Intercept) 612.10 24.741
Days 35.07 5.922 0.07
Residual 654.94 25.592
Number of obs: 180, groups: Subject, 18
Fixed effects:
Estimate Std. Error t value
(Intercept) 251.405 6.825 36.838
Days 10.467 1.546 6.771
Correlation of Fixed Effects:
(Intr)
Days -0.138ばらつきの割合を見る
共分散行列の対角要素に切片や変数の分散が入っています。 また、残差の標準偏差はsigmaという名前で入っているので、それを二乗することで残差の分散を取り出します。
var_intercept <- result$varcor$Subject[1, 1] # 切片の分散 variance
var_days <- result$varcor$Subject[2, 2] # Daysのvariance
var_residual <- result$sigma^2 # 残差のvariance
var_sum <- sum(var_intercept, var_days, var_residual)
ratio_var_intercept <- var_intercept / var_sum
ratio_var_days <- var_days / var_sum
ratio_var_residual <- var_residual / var_sum切片、傾き、残差のばらつきがそれぞれ得られます。
予測線を描く
predict関数に作成したモデルを入れることで、簡単にそれぞれの点の予測値を計算してくれます。
plot(sleepstudy$Days, sleepstudy$Reaction, col = sleepstudy$Subject, xlab = "Days", ylab = "Reaction Time (ms)", yaxt = "n", main = "Original")
axis(2, las = 2)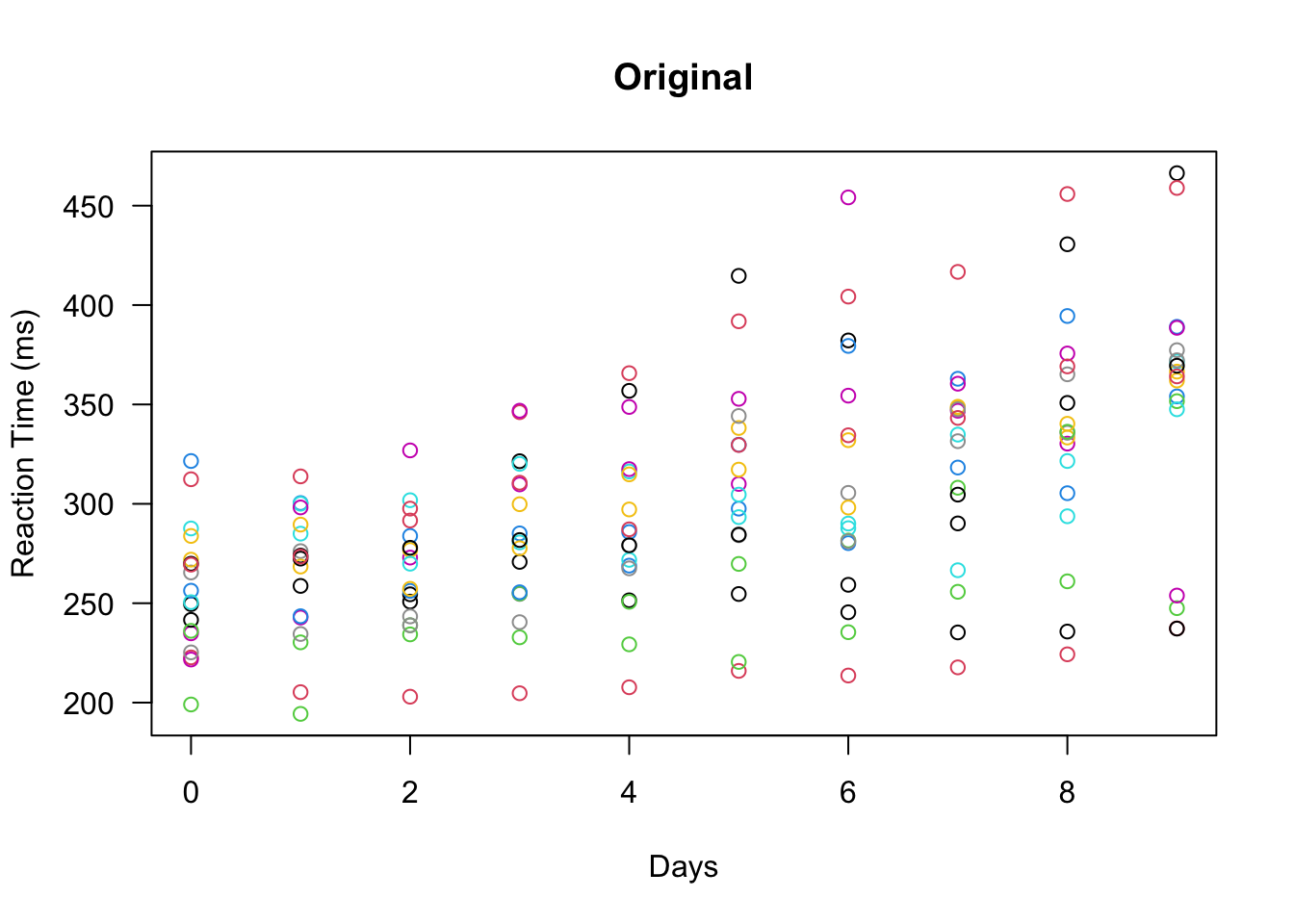
predictions <- predict(model)
plot(sleepstudy$Days, predictions, col = sleepstudy$Subject, xlab = "Days", ylab = "Reaction Time (ms)", yaxt = "n", main = "Prediction")
axis(2, las = 2)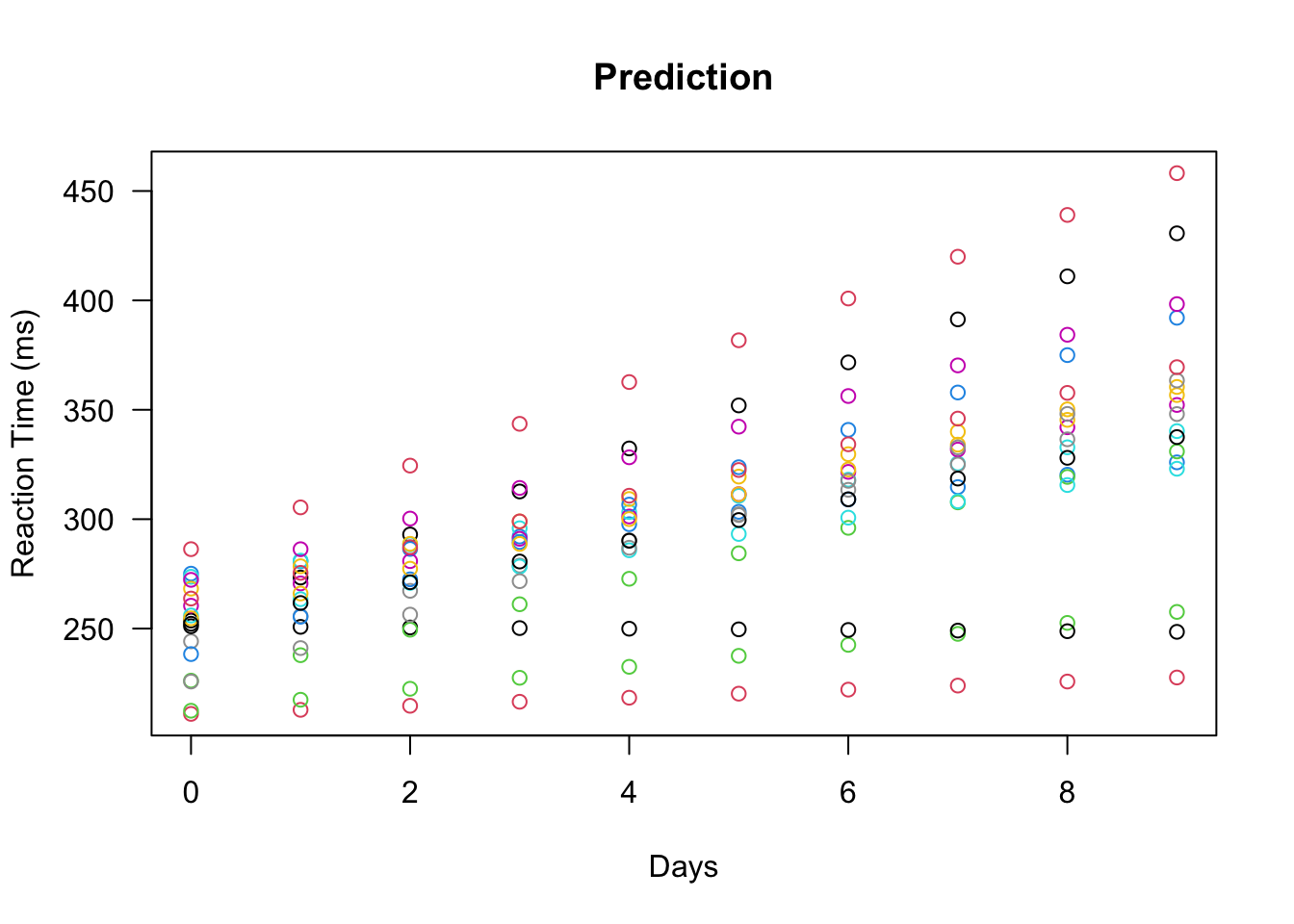
残差について
summary(model)で得られたresultオブジェクトにはresidualsという名前で残差が入っています。 しかし、これはそのままの残差ではなく、スケール残差 scaled residualsというものになります。 scaled residualsは以下の式で計算されます。
\[ \mathrm{scaled \ residuals} = \frac{\mathrm{residuals}}{\mathrm{SD(residuals)}} \]
SDは標準偏差 standard deviationです。 redisuals自体は、実際のデータ - 予測値で得られます。 予測値自体は先ほどpredict(model)で得られた値です。
「実測データ-予測値」とモデルから得られる残差が一致することを確かめます。
1 2 3 4 5 6
-4.1036558 -14.6252175 -42.1955792 8.7773590 24.5231973 62.6951356
7 8 9 10 11 12
10.5425739 -101.1788879 19.5915504 35.6934887 11.7275332 -7.5881721
13 14 15 16 17 18
-11.7237775 -11.8421828 -10.6806881 -4.2825935 -8.4616988 -6.2124042
19 20 21 22 23 24
-1.4915095 9.6793851 -13.3907959 -23.1309254 11.8384451 5.3416156
25 26 27 28 29 30
-3.2110139 -17.0789433 -7.1344728 8.1773977 8.4203682 -10.0952613
31 32 33 34 35 36
46.4468756 19.6515399 -2.5450957 -6.9215313 -11.9101670 -5.7749026
37 38 39 40 41 42
-28.7737382 3.5950261 -14.9697095 28.0765548 13.9424826 3.9372082
43 44 45 46 47 48
13.3604339 24.2577595 13.0223852 -17.3335892 -27.9746635 9.3706621
49 50 51 52 53 54
-39.0975122 31.3393134 -25.5840727 -27.8279817 -7.8735907 18.7388004
55 56 57 58 59 60
16.2377914 -1.4226176 132.5465735 15.0206645 -11.7052445 -98.3362534
61 62 63 64 65 66
15.5967865 11.0657367 -11.9636132 0.8331369 -12.0492130 18.7026372
67 68 69 70 71 72
2.3189873 8.8887374 -16.8348125 1.6043376 21.3006103 20.4868426
73 74 75 76 77 78
-23.8915250 -24.1257926 -11.3155602 -17.6906278 -7.8988954 6.5573370
79 80 81 82 83 84
-0.7605306 29.2497018 -9.4631364 23.1606428 3.9890220 20.5853012
85 86 87 88 89 90
1.5199804 4.9891597 -3.9098611 -13.7662819 -13.0383027 -11.2609235
91 92 93 94 95 96
26.0710083 8.4146572 -32.8754939 2.5399550 3.0546039 10.0651527
97 98 99 100 101 102
3.3912016 -3.2721495 16.8042994 0.7611483 9.9083238 -7.5188944
103 104 105 106 107 108
-10.5507125 -6.1950306 -22.0474488 -14.6240669 -14.4743850 0.4220968
109 110 111 112 113 114
16.9599787 20.6837606 17.9617293 -11.9622745 -16.2934782 -34.0524820
115 116 117 118 119 120
-37.7445858 5.9821105 38.6204067 5.0128029 19.5000992 -3.0159046
121 122 123 124 125 126
-5.4564690 36.6226071 -0.9931168 2.2500593 -13.9636645 11.3905116
127 128 129 130 131 132
-12.9485123 -41.5516362 5.9426399 24.5143161 -50.5916830 11.9218299
133 134 135 136 137 138
26.6031428 32.5768557 20.4582686 10.5434816 -1.8619055 -9.8591926
139 140 141 142 143 144
-8.6544797 -9.7566668 17.2429295 2.4168287 -20.1171721 -11.0424730
145 146 147 148 149 150
14.7836262 5.8354254 -24.5822755 14.0658237 -5.1165771 9.7770220
151 152 153 154 155 156
-0.5281055 -6.5583765 -17.4708474 -31.1884183 -19.4138892 41.9527399
157 158 159 160 161 162
-36.3826310 14.7649981 17.0527272 8.8287562 17.6682491 10.7515193
163 164 165 166 167 168
6.7284896 1.1399599 -10.9581699 -15.0957996 -49.8211294 -13.9354591
169 170 171 172 173 174
22.7355112 31.9448814 5.6920030 -1.9970050 10.3744870 11.6579789
175 176 177 178 179 180
-23.5523291 7.1313629 0.2542548 -2.7589532 11.4115388 -5.3578693 1 2 3 4 5 6
-4.1036558 -14.6252175 -42.1955792 8.7773590 24.5231973 62.6951356
7 8 9 10 11 12
10.5425739 -101.1788879 19.5915504 35.6934887 11.7275332 -7.5881721
13 14 15 16 17 18
-11.7237775 -11.8421828 -10.6806881 -4.2825935 -8.4616988 -6.2124042
19 20 21 22 23 24
-1.4915095 9.6793851 -13.3907959 -23.1309254 11.8384451 5.3416156
25 26 27 28 29 30
-3.2110139 -17.0789433 -7.1344728 8.1773977 8.4203682 -10.0952613
31 32 33 34 35 36
46.4468756 19.6515399 -2.5450957 -6.9215313 -11.9101670 -5.7749026
37 38 39 40 41 42
-28.7737382 3.5950261 -14.9697095 28.0765548 13.9424826 3.9372082
43 44 45 46 47 48
13.3604339 24.2577595 13.0223852 -17.3335892 -27.9746635 9.3706621
49 50 51 52 53 54
-39.0975122 31.3393134 -25.5840727 -27.8279817 -7.8735907 18.7388004
55 56 57 58 59 60
16.2377914 -1.4226176 132.5465735 15.0206645 -11.7052445 -98.3362534
61 62 63 64 65 66
15.5967865 11.0657367 -11.9636132 0.8331369 -12.0492130 18.7026372
67 68 69 70 71 72
2.3189873 8.8887374 -16.8348125 1.6043376 21.3006103 20.4868426
73 74 75 76 77 78
-23.8915250 -24.1257926 -11.3155602 -17.6906278 -7.8988954 6.5573370
79 80 81 82 83 84
-0.7605306 29.2497018 -9.4631364 23.1606428 3.9890220 20.5853012
85 86 87 88 89 90
1.5199804 4.9891597 -3.9098611 -13.7662819 -13.0383027 -11.2609235
91 92 93 94 95 96
26.0710083 8.4146572 -32.8754939 2.5399550 3.0546039 10.0651527
97 98 99 100 101 102
3.3912016 -3.2721495 16.8042994 0.7611483 9.9083238 -7.5188944
103 104 105 106 107 108
-10.5507125 -6.1950306 -22.0474488 -14.6240669 -14.4743850 0.4220968
109 110 111 112 113 114
16.9599787 20.6837606 17.9617293 -11.9622745 -16.2934782 -34.0524820
115 116 117 118 119 120
-37.7445858 5.9821105 38.6204067 5.0128029 19.5000992 -3.0159046
121 122 123 124 125 126
-5.4564690 36.6226071 -0.9931168 2.2500593 -13.9636645 11.3905116
127 128 129 130 131 132
-12.9485123 -41.5516362 5.9426399 24.5143161 -50.5916830 11.9218299
133 134 135 136 137 138
26.6031428 32.5768557 20.4582686 10.5434816 -1.8619055 -9.8591926
139 140 141 142 143 144
-8.6544797 -9.7566668 17.2429295 2.4168287 -20.1171721 -11.0424730
145 146 147 148 149 150
14.7836262 5.8354254 -24.5822755 14.0658237 -5.1165771 9.7770220
151 152 153 154 155 156
-0.5281055 -6.5583765 -17.4708474 -31.1884183 -19.4138892 41.9527399
157 158 159 160 161 162
-36.3826310 14.7649981 17.0527272 8.8287562 17.6682491 10.7515193
163 164 165 166 167 168
6.7284896 1.1399599 -10.9581699 -15.0957996 -49.8211294 -13.9354591
169 170 171 172 173 174
22.7355112 31.9448814 5.6920030 -1.9970050 10.3744870 11.6579789
175 176 177 178 179 180
-23.5523291 7.1313629 0.2542548 -2.7589532 11.4115388 -5.3578693 一致していますね。
残差の可視化
残差を可視化してみます。 わかりやすさのために、最初の10点について見てみます。 ここでは、prediction(model)で得られた値でなく、モデルから得られる残差でプロットします。
residuals <- result$residuals * result$sigma
plot(sleepstudy$Days[1:10], sleepstudy$Reaction[1:10], col = 1, xlab = "Days", ylab = "Reaction Time (ms)", yaxt = "n")
axis(2, las = 2)
points(sleepstudy$Days[1:10], sleepstudy$Reaction[1:10] - residuals[1:10], col = 2)
segments(x0 = sleepstudy$Days[1:10], y0 = sleepstudy$Reaction[1:10], x1 = sleepstudy$Days[1:10], y1 = sleepstudy$Reaction[1:10] - residuals[1:10], col = 3)
legend("topleft", legend = c("Original", "Prediction", "Residuals"), col = 1:3, pch = c(1, 1, NA), lty = c(NA, NA, 1))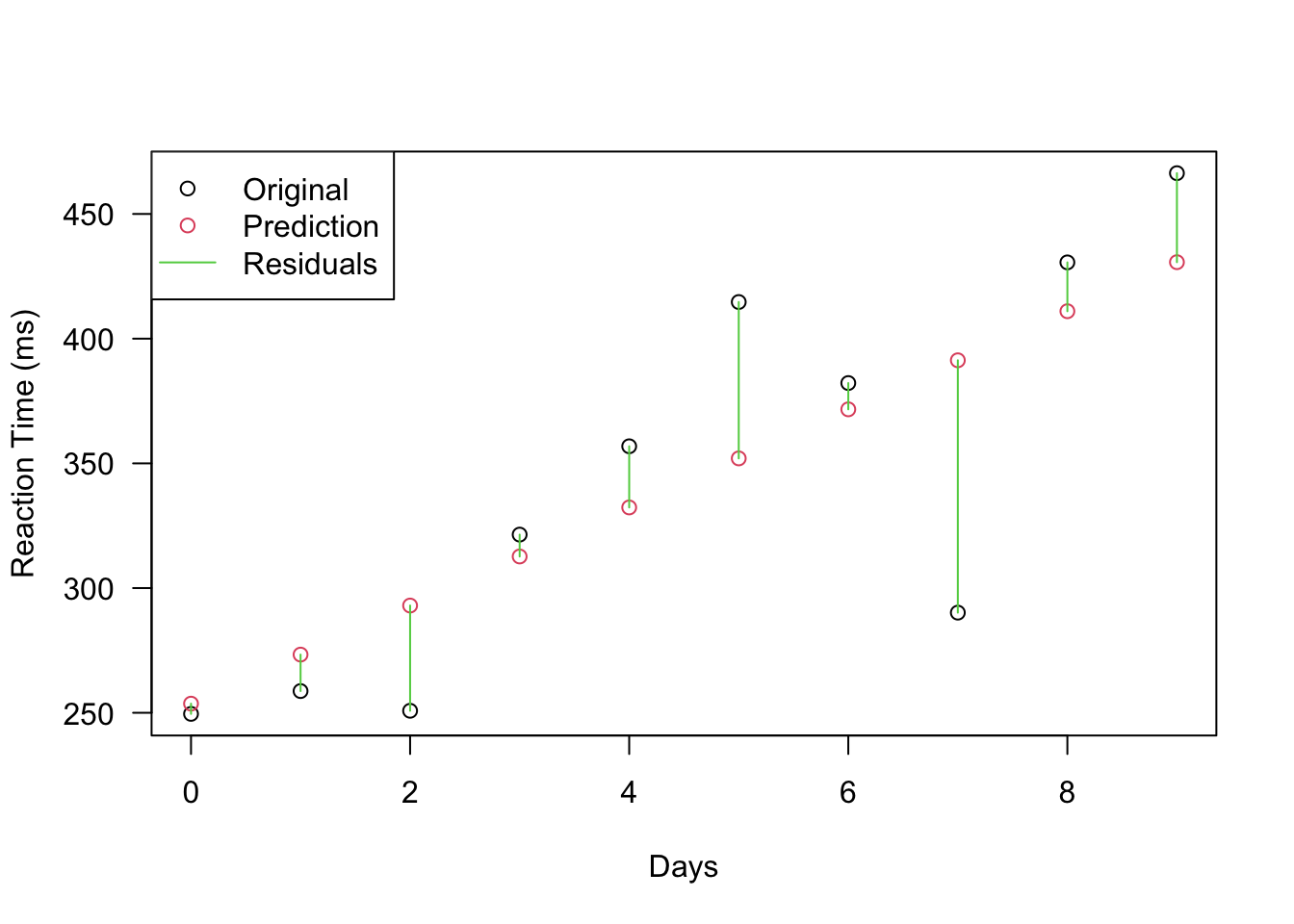
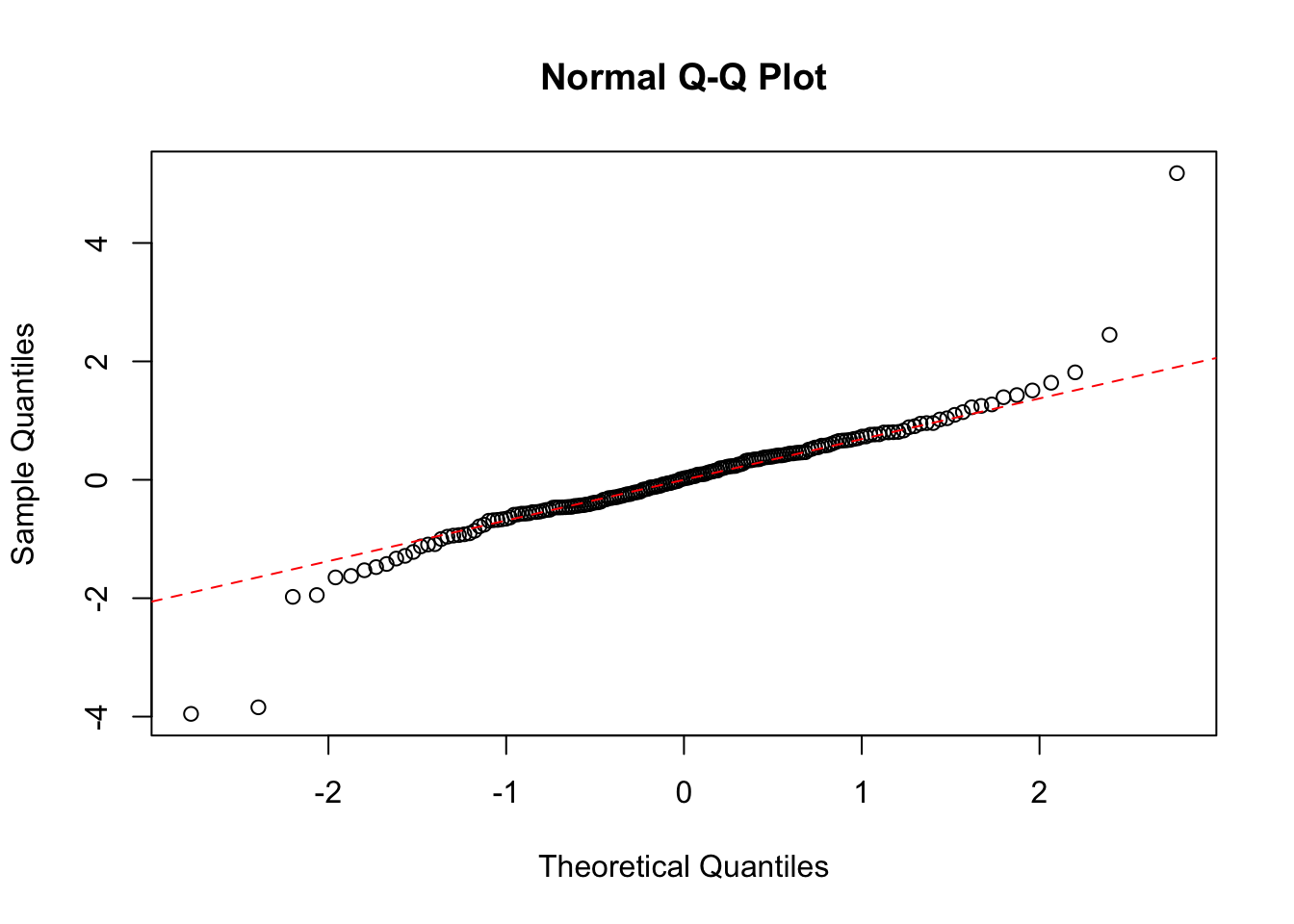
残差の正規性を確認する
Q-Qプロットを作成するには、qqnormとqqlineが有用です。 Q-Qプロットの意味については今回は掘り下げません。